Nature | 克隆拷貝數多樣性影響肺癌生存
關鍵詞
肺癌、克隆拷貝數、ALPACA方法、染色體不穩定性、生存預后
摘要總結
這篇文章是2025年8月發表在《Nature》雜志上的一篇研究,標題為“Clone copy number diversity is linked to survival in lung cancer”。這篇文章通過開發新型算法ALPACA,探索了肺癌中單核苷酸變異(SNV)和體細胞拷貝數變異(SCNA)的共同演化,這對于揭示肺癌轉移機制、提升預后預測具有重要意義。
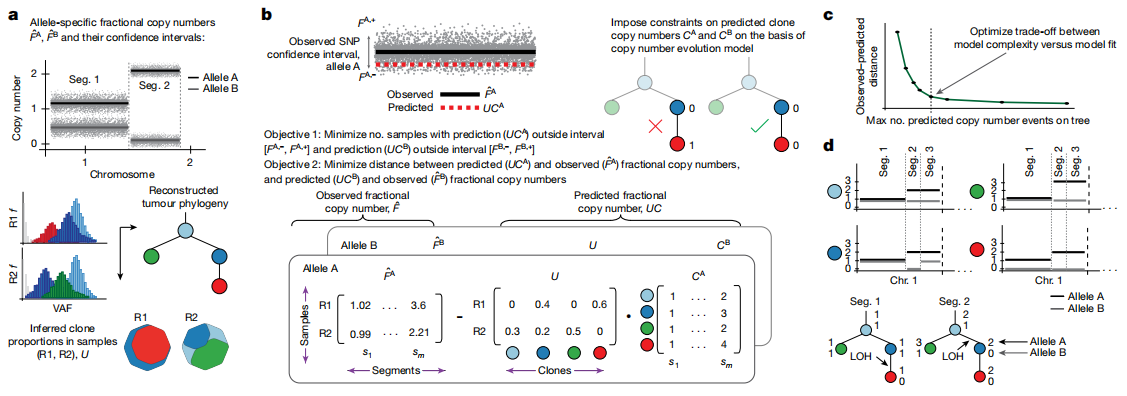
研究背景
癌癥發展過程中,SNV和SCNA共同驅動克隆演化,導致腫瘤異質性和染色體不穩定性(CIN)。現有方法難以從bulk DNA測序數據中準確推斷克隆特異性拷貝數,導致對腫瘤多樣性和轉移機制的理解不足。TRACERx421研究提供了多樣本肺癌數據,但缺乏整合SNV和SCNA的工具。本研究旨在填補這一空白,探討克隆拷貝數多樣性與肺癌生存的相關性。
TRACERx421,全稱是 “Tracking Cancer Evolution through Therapy (Rx)”(通過治療追蹤癌癥演化),涉及 421 名未經治療的非小細胞肺癌(NSCLC)患者。TRACERx421 的目標是揭示癌癥克隆演化、轉移機制以及與預后相關的基因組特征,為精準醫學提供數據支持。
研究方法
研究開發了ALPACA算法,利用SNV頻率重建的腫瘤系統樹作為框架,結合多樣本bulk DNA測序數據推斷克隆特異性等位基因拷貝數。輸入包括腫瘤克隆樹、克隆比例和等位基因分數拷貝數,通過混合整數線性規劃和模型選擇優化SCNA演化。基準測試使用MASCoTE和Tx模擬腫瘤(n=150),并與HATCHet2、cloneHD等比較。應用到TRACERx421的395個原發肺癌和126個轉移腫瘤,分析SCNA模式、事件時序和轉移相關性,使用Bradley-Terry模型排序事件,并通過混合效應模型評估拷貝數變化。
研究結果
-
ALPACA在模擬腫瘤中準確率高于現有方法(P<0.001)
ALPACA是一種新算法,用于從多樣本bulk DNA測序數據中推斷腫瘤克隆特異性拷貝數(包括單核苷酸變異SNV和體細胞拷貝數變異SCNA)。在模擬腫瘤數據中,ALPACA的預測準確率顯著優于現有方法(如HATCHet2和cloneHD),統計顯著性P值小于0.001,表明結果高度可靠。 -
ALPACA可發現轉移播種克隆中的LOH和擴增事件(如BRCA2、RB1損失和CCND1擴增)
ALPACA能夠識別轉移過程中“播種克隆”(即引發轉移的克隆)中的特定基因組改變,包括“失雙等位基因”(Loss of Heterozygosity, LOH)和基因擴增。具體例子包括BRCA2、RB1(腫瘤抑制基因)的LOH以及CCND1(細胞周期調控基因)的擴增。 -
肺腺癌(LUAD)中臂級擴增早于焦點擴增,驅動SNV(如EGFR、KRAS)較早發生;肺鱗癌(LUSC)中臂級事件多為樹干
研究分析了LUAD和LUSC兩種肺癌亞型中基因組事件的演化順序。LUAD中,染色體臂級擴增(影響較大染色體區域)先于焦點擴增(影響特定基因),而驅動性SNV(如EGFR、KRAS突變)發生在早期。LUSC中,臂級事件(如LOH或擴增)多發生在“樹干”(即所有克隆共有的早期祖先克隆)。 -
轉移克隆顯示更高SCNA速率(P=0.0474)
使用線性混合效應模型分析TRACERx421數據,發現轉移克隆在每條系統發育樹邊上的SCNA數量顯著增加。這可能反映轉移克隆在適應新微環境中需要更多基因組改變。 -
播種克隆中TSG損失和CCND1擴增富集
引發轉移的播種克隆中,腫瘤抑制基因(TSG,如SMARCA4、KEAP1)的LOH以及CCND1擴增事件顯著富集,表明TSG損失可能削弱腫瘤抑制,CCND1擴增促進細胞周期加速,為轉移提供遺傳優勢。 -
克隆拷貝數多樣性增加與肺癌患者無病生存期縮短相關
腫瘤中克隆拷貝數多樣性越高(即不同克隆間拷貝數差異越大),肺癌患者的無病生存期(疾病未復發的時間)越短。
文章的亮點和局限
亮點:ALPACA創新性地整合SNV和SCNA,提供克隆級分辨率,揭示隱藏在minor克隆中的事件,并關聯轉移和預后,有助于精準醫學。
局限:依賴高SNV突變率(如>0.3/細胞分裂),可能不適用于低突變腫瘤;基于bulk數據,可能忽略罕見克隆;模擬和真實數據驗證有限,需更多隊列確認。
公開數據
The TRACERx primary and matched primary–metastasis processed data have been deposited at Zenodo at https://doi.org/10.5281/zenodo.7822002 (ref.?54). The single-cell study from the TRACERx PEACE study has been deposited at Zenodo at https://doi.org/10.5281/zenodo.13754279 (ref.?55). Processed data used in this publication have been deposited at Zenodo at https://doi.org/10.5281/zenodo.15519765 (ref.?56). The WES data (from the TRACERx study) used during this study have been deposited at the European Genome–Phenome Archive (EGA), which is hosted by the European Bioinformatics Institute and the Centre for Genomic Regulation, under the accession code EGAS00001006494; raw single-cell DNA-sequencing data used in this study from the patients enrolled in the TRACERx and PEACE studies have been deposited at the EGA under the accession code EGAD00001015411.
本文來自博客園,作者:橙子牛奶糖(陳文燕),轉載請注明原文鏈接:http://www.rzrgm.cn/chenwenyan/p/19070880



 浙公網安備 33010602011771號
浙公網安備 33010602011771號