使用kraken2 命令對重測序數據進行物種分類
001、
## step1: mkdir sheep_db ## 創建數據庫目錄 ## step2: download the taxonomy information kraken2-build --download-taxonomy --db sheep_db ## 下載物種分類信息 ## step3: download the human genome kraken2-build --download-library human --db sheep_db ## 下載人類基因組 ## step4: download the virus genome kraken2-build --download-library viral --db sheep_db ## 下載病毒基因組 ## step5: download the bacteria genome kraken2-build --download-library bacteria --db sheep_db ## 下載細菌基因組 ## step6: add the sheep genome kraken2-build --add-to-library /public/home/b20223040323/software/kraken2-2.1.3/sheep_ref/GCF_016772045.2_ARS-UI_Ramb_v3.0_genomic.fna --db sheep_db ## 添加綿羊基因組 ## step7: build database kraken2-build --build --db sheep_db --threads 8 ## 構建數據庫 ## step8: kraken2 --db /public/home/b20223040323/software/kraken2-2.1.3/sheep_db --paired TUJ7_trim_1P.fastq.gz TUJ7_trim_2P.fastq.gz --report s_wrong.kraken.report --output s_wrong.kraken.out --use-names --threads 16 ## 運算
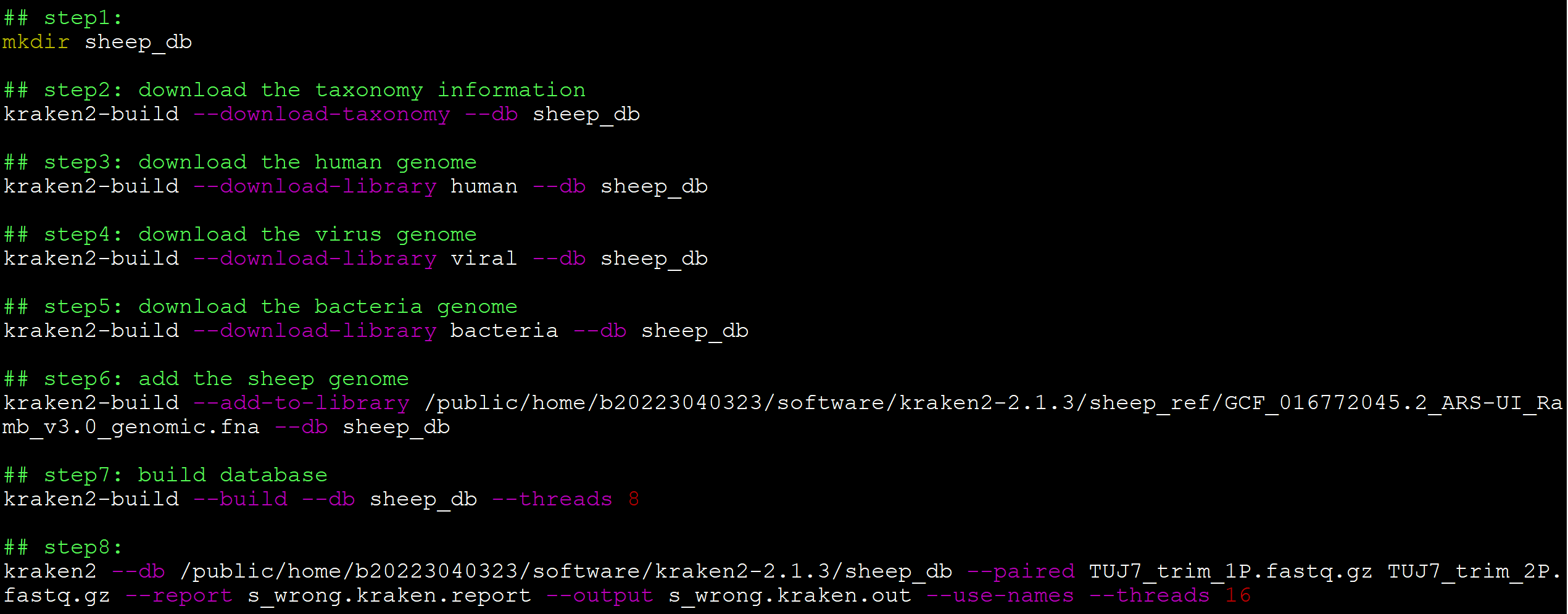
。
002、 提取目標物種的reads
awk -F "\t" '$3 == "Ovis aries (taxid 9940)" {print $2}' sample_name.kraken.out > sample_name.sheep_reads ## 提取reads 的 ID seqtk subseq sample_name_trim_1P.fastq.gz sample_name.sheep_reads | gzip > sample_name_F_trim_1P.fastq.gz ## 提取read1 seqtk subseq sample_name_trim_2P.fastq.gz sample_name.sheep_reads | gzip > sample_name_F_trim_2P.fastq.gz ## 提取read2
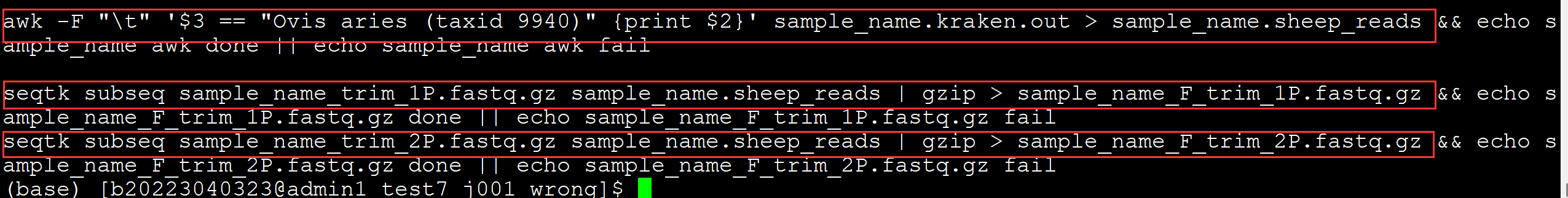
。



 浙公網安備 33010602011771號
浙公網安備 33010602011771號