易基因:常見生信分析圖細節解析(第一期)|生信解析
大家好,這里是專注表觀組學十余年,領跑多組學科研服務的易基因。
生信分析圖作為基因組、轉錄組等組學研究結果的可視化核心,常見類型包括火山圖(Volcano Plot)、Deeptools信號熱圖、IGV的動態變化、PLS-DA圖、相關性圖、tSNE圖、UMAP圖、柱狀堆疊圖、山巒圖等。這些圖表通過不同形式展現差異基因、染色質開放位點、功能富集等關鍵信息。對于剛入門的研究者來說,理解這些圖表背后的生物學信息可能頗具挑戰。今天,易基因將帶您深入解析高分文章中常見的生信分析圖,幫助您掌握它們的應用場景和具體含義。
1、火山圖
標題:Defining the KRAS- and ERK-dependent transcriptome in KRAS-mutant cancers(揭示KRAS突變癌癥中的KRAS和ERK依賴性轉錄組)
發表時間:2024年6月7日
發表期刊:Science(火山圖)
影響因子:IF45.8/Q1
DOI:10.1126/science.adk0775
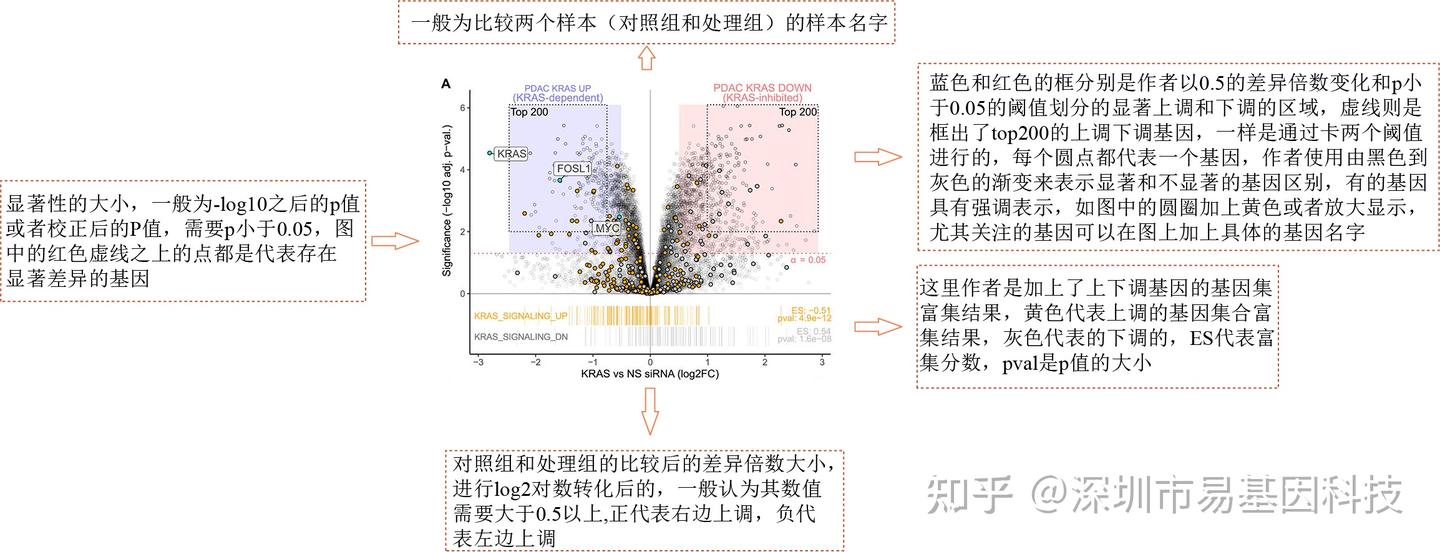
Figure 1 KRAS-dependent gene expression changes upon acute (24 hours) KRAS suppression in eight KRAS-mutant PDAC cell lines transiently transfected with KRAS or control non-specific (NS) siRNA.
圖1在八種KRAS突變型PDAC細胞系中,經KRAS或對照非特異性(NS)siRNA瞬時轉染后,急性(24小時)KRAS抑制導致的KRAS依賴性基因表達變化。
易小結
火山圖(Volcano plot)將統計術語中的顯著性(-log10 P-value)和差異變化倍數(Fold change)相結合,直觀地識別對照組和處理組中變化幅度較大且具有統計學意義的單個數據點(基因、蛋白及代謝物等)。常應用于研究基因組、表觀遺傳、轉錄組、代謝組和蛋白質組等數據分析。
2、柱狀堆疊圖
標題:A Gram-negative-selective antibiotic that spares the gut microbiome(一種選擇性針對革蘭氏陰性菌且不傷害腸道微生物組的抗生素)
發表時間:2024年5月29日
發表期刊:Nature(柱狀堆疊圖)
影響因子:IF48.5/Q1
DOI:10.1038/s41586-024-07502-0
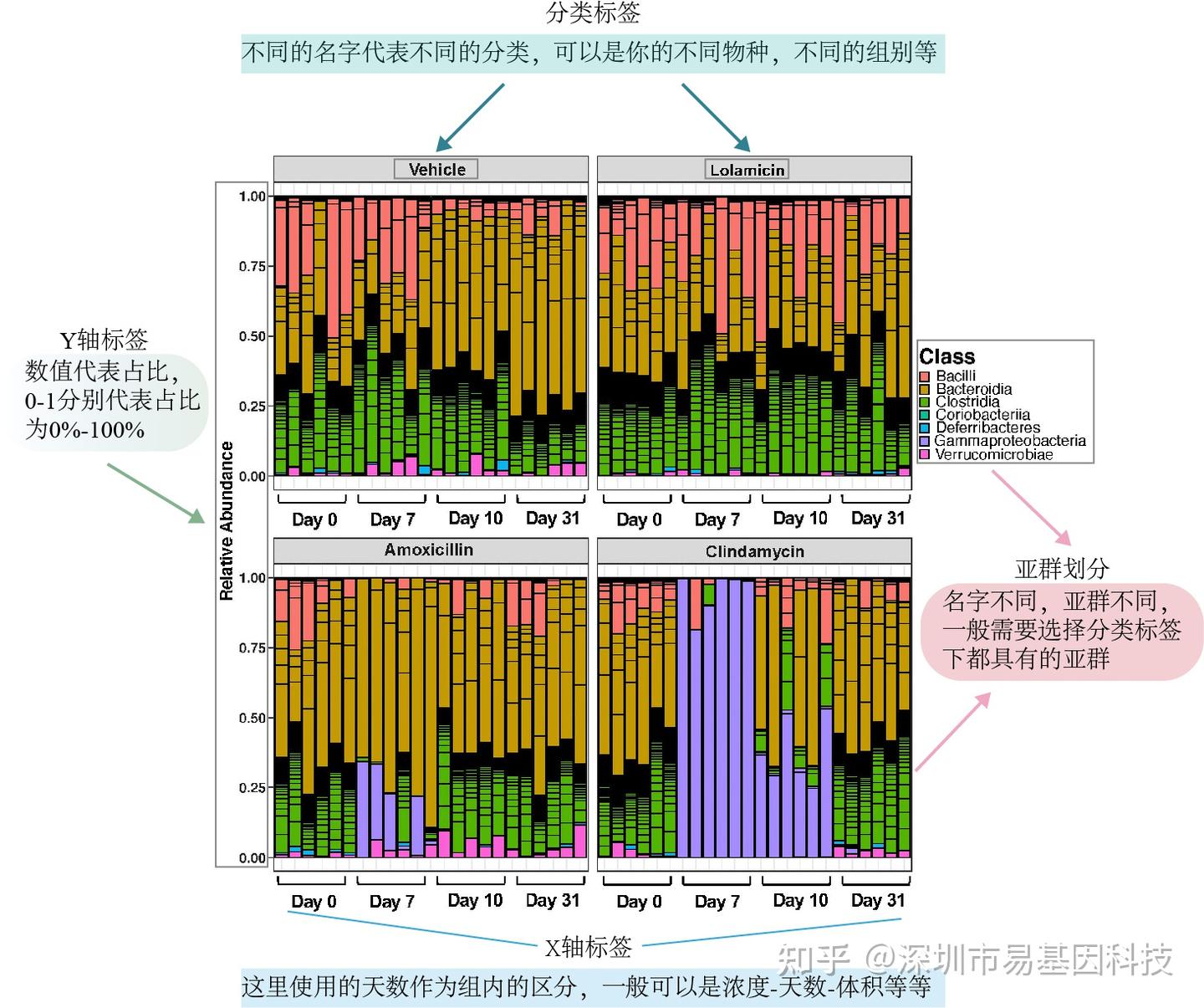
Figure 2 Bacterial composition of mouse fecal microbiota obtained by full-length 16s rRNA sequencing at the Class level.
圖2通過全長16s rRNA測序在綱水平上獲得的小鼠糞便微生物群細菌組成。
易小結
堆疊柱狀圖(Stacked bar chart)多維度展示不同樣本之間的動態變化趨勢,還可以探究哪一部分比例(細菌數量、基因數量等)最大,以及每一部分的變動情況(從有到無及從無到有)。常應用于研究基因組、微生物組、表觀遺傳學等。
3、山巒圖
標題:An atlas of epithelial cell states and plasticity in lung adenocarcinoma(肺腺癌上皮細胞狀態與可塑性的圖譜)
發表時間:2024年2月28日
發表期刊:Nature(山巒圖)
影響因子:IF48.5/Q1
DOI:10.1038/s41586-024-07113-9
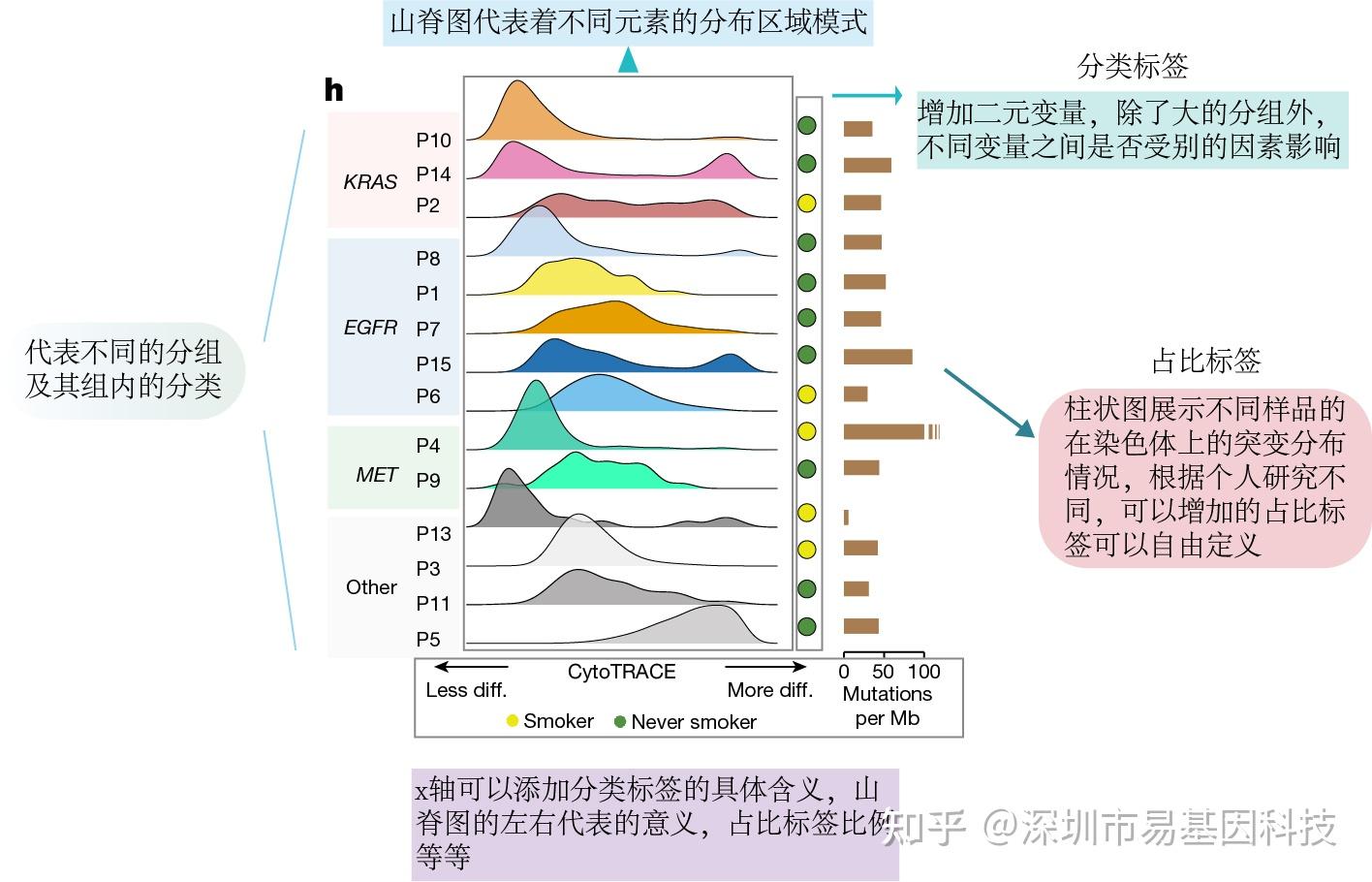
Figure 3 Per sample distribution of malignant cell CytoTRACE scores.
圖3每個樣本中惡性細胞CytoTRACE評分的分布。
易小結
山巒圖(Ridge plot)直觀展示樣本內部不同處理組或者時間維度內相同元素(如某基因、某蛋白及活性分數等)動態變化趨勢。常應用于研究表觀遺傳學、微生物組、代謝組等。
4、GSEA富集分析圖
標題:B-cell specific checkpoint molecules that regulate anti-tumor immunity(調控抗腫瘤免疫的B細胞特異性檢查點分子)
發表時間:2023年6月21日
發表期刊:Nature(GSEA富集分析圖)
影響因子:IF48.5/Q1
DOI:10.1038/s41586-023-06231-0
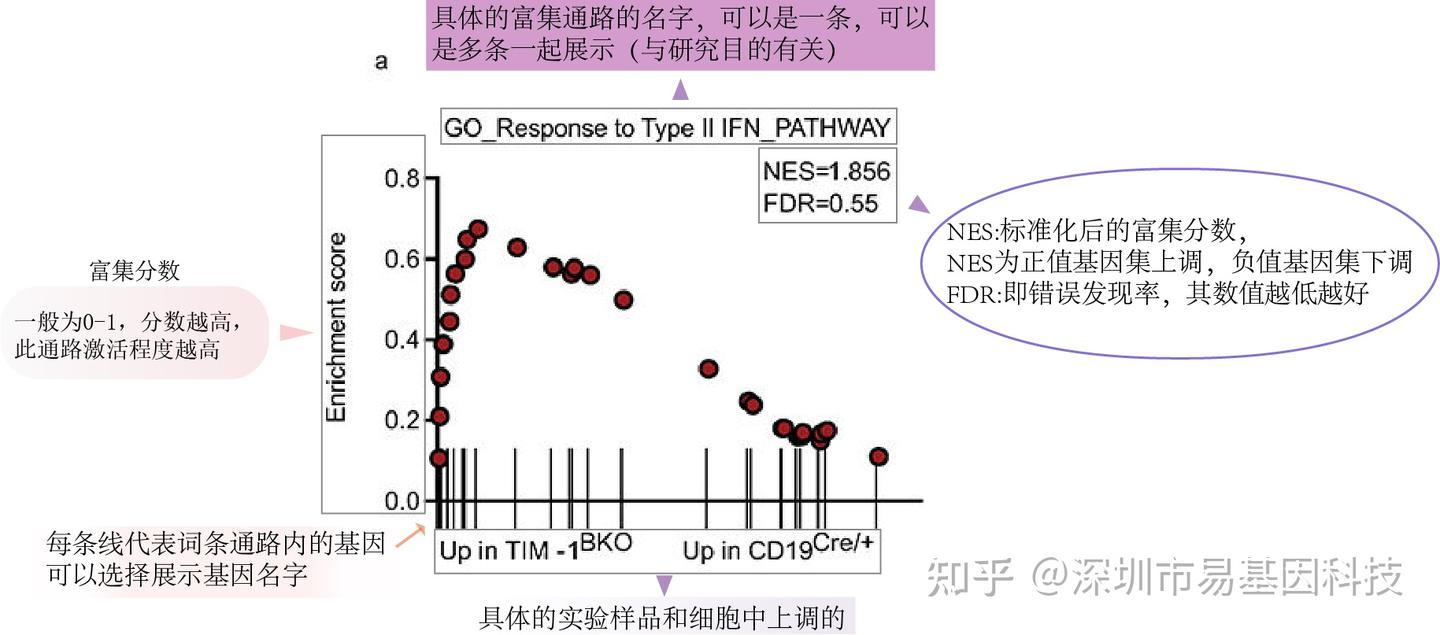
Figure 4 GSEA analysis for the “Response to type II IFN pathway” of tumor-infiltrating TIM-1BKO and CD19Cre/+ B cells.
圖4 TIM-1BKO和CD19Cre/+ B細胞腫瘤浸潤中"II型干擾素反應通路"的GSEA分析。
易小結
GSEA富集分析是基因集富集分析,用來確定一組先驗定義的基因集是否在兩種生物狀態之間顯示出統計學上顯著的、一致的差異,相較于傳統的基因功能富集,其主要提供感興趣的通路在你的樣本中的實際激活情況,可以是正激活和負激活兩類。常用于表觀遺傳學、基因組等研究中。
5、deeptools熱圖
標題:c-JUN: a chromatin repressor that limits mesoderm differentiation in human pluripotent stem cells(c-JUN:一種染色質阻遏蛋白,可限制人多能干細胞中胚層分化)
發表時間:2025年1月29日
發表期刊:Nucleic Acids Research(deeptools熱圖)
影響因子:IF13.1/Q1
DOI:10.1093/nar/gkaf001
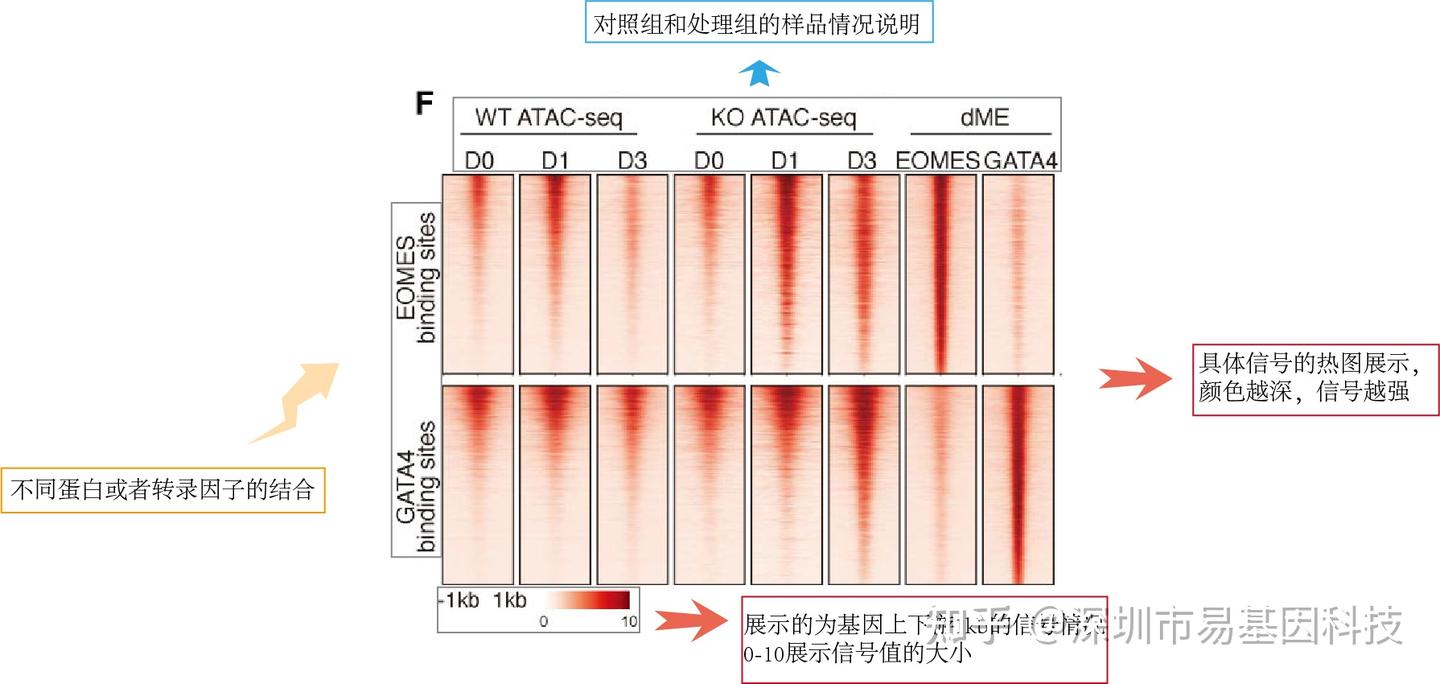
Figure 5 Heatmap shows the signal of WT and c-JUN KO ATAC-seq data during hPSC (D0) differentiated into mesoderm cell (D3), and EOMES, GATA4 ChIP-seq data in dME on EOMES and GATA4 binding sites.
圖5 熱圖顯示了WT和c-JUN KO ATAC-seq數據在hPSC(D0)分化為中胚層細胞(D3)期間的信號,以及EOMES、GATA4在dME中對EOMES和GATA4結合位點的ChIP-seq數據。
易小結
Deeptools熱圖信號分析是多樣本、多技術之間不同區域的信號值直觀展示,可以用于研究ChIP-seq、ATAC-seq、CUT&RUN及CUT&Tag等染色質結構與開放等信號值,常用于研究轉錄因子或組蛋白修飾在不同樣本之間結合的變化、染色質開放區域的動態變化等研究。
6、IGV信號圖
標題:c-JUN: a chromatin repressor that limits mesoderm differentiation in human pluripotent stem cells(c-JUN:一種染色質阻遏蛋白,可限制人多能干細胞中胚層分化)
發表時間:2025年1月29日
發表期刊:Nucleic Acids Research(IGV信號圖)
影響因子:IF13.1/Q1
DOI:10.1093/nar/gkaf001
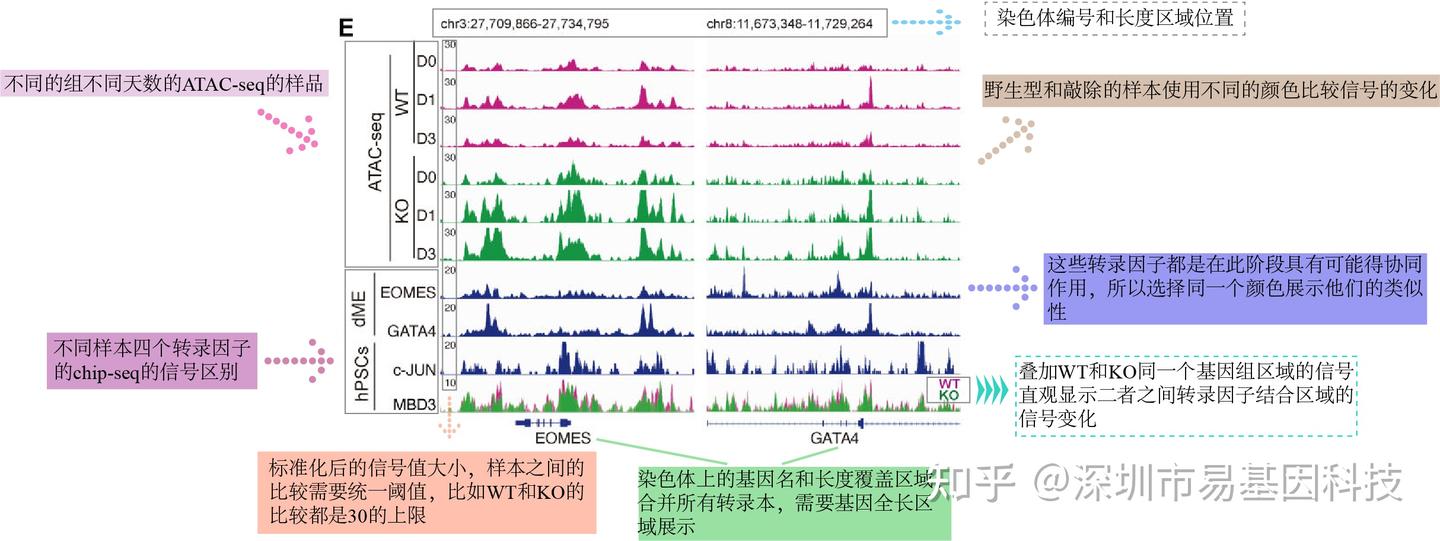
Figure 6 Genome view of the ATAC-seq data during hPSC (D0) differentiated into mesoderm cell (D3), and EOMES (GSM1505630, GSM1505631) , GATA4 (GSM1505644, GSM1505645) ChIP-seq data in dME and c-JUN, MBD CUT&RUN data in hPSC on select target genes.
圖6 ATAC-seq數據在hPSC(D0)分化為中胚層細胞(D3)過程中的基因組視圖,以及EOMES(GSM1505630, GSM1505631)、GATA4(GSM1505644, GSM1505645)在dME中的ChIP-seq數據,和c-JUN、MBD在hPSC中對選定靶基因的CUT&RUN數據。
易小結
IGV(Integrative Genomics Viewer)信號分析是展示單個或多個基因不同樣本、相同技術之間基因組區域的信號值動態變化,可以用于研究ChIP-seq、ATAC-seq、CUT&RUN及CUT&Tag等染色質結構與開放等信號值,常用于研究轉錄因子或組蛋白修飾在不同樣本之間結合的變化、染色質開放區域的動態變化等研究。
7、UMAP
標題:Deciphering the regulatory networks of human male germline development from embryo to adulthood(從胚胎到成年的人類雄性生殖細胞發育調控網絡解析)
發表時間:2025年5月21日
發表期刊:Biochimica et Biophysica Acta-Molecular Basis of Disease(UMAP)
影響因子:IF4.2/Q1
DOI:10.1016/j.bbadis.2025.167918
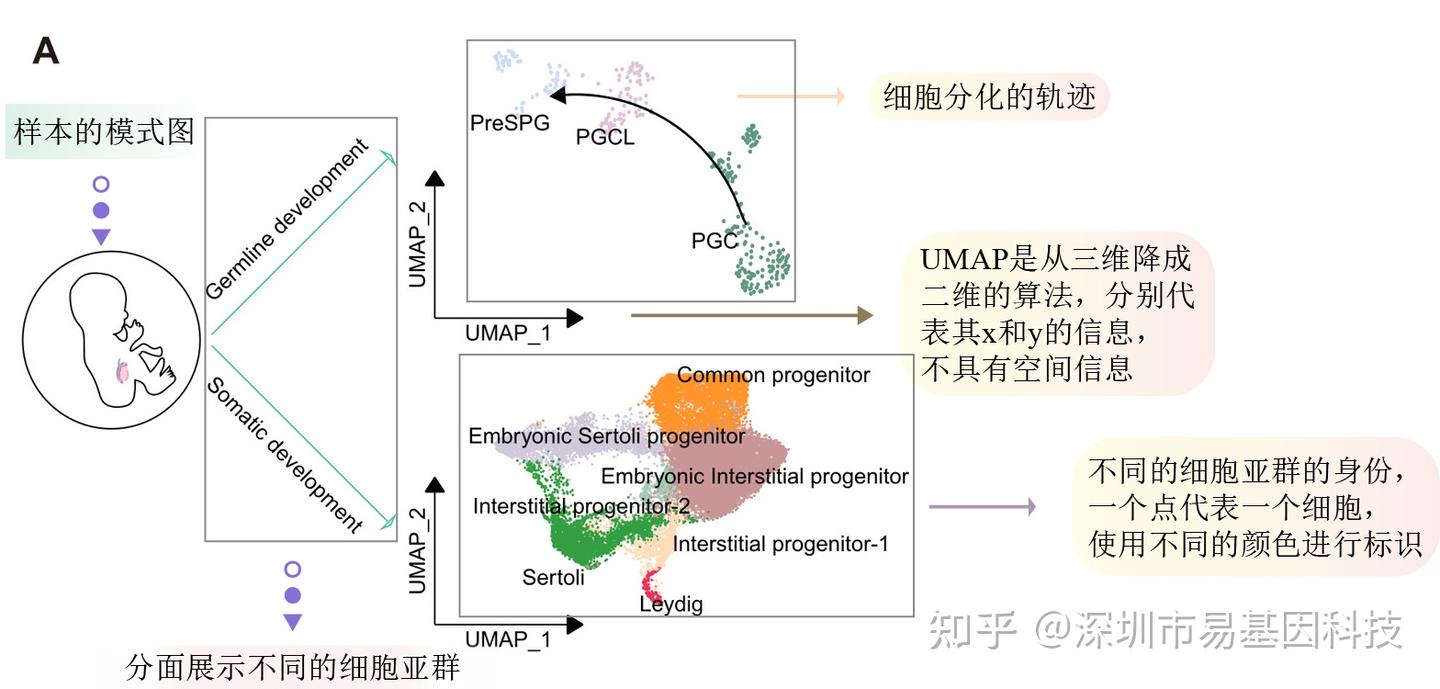
Figure 7 Developmental lineages of germ cells and main somatic cells during the embryonic period.
圖7胚胎時期生殖細胞與主要體細胞的發育譜系。
易小結
UMAP(Uniform manifold approximation and projection)降維分析即學習高維空間中的流形結構,找到該流形的低維表示,用于數據可視化和分析細胞(單細胞轉錄組和空間轉錄組等轉錄組分析),常用于展示細胞亞群構成、細胞發育時期動態變化、不同樣本之間的主要細胞亞群構成的變化、稀有亞群的分布等等。
8、PLS-DA圖
標題:Mapping pesticide-induced metabolic alterations in human gut bacteria(繪制農藥引起的人類腸道細菌代謝變化圖譜)
發表時間:2025年5月10日
發表期刊:Nature Communications(PLS-DA圖)
影響因子:IF15.7/Q1
DOI:10.1038/s41467-025-59747-6
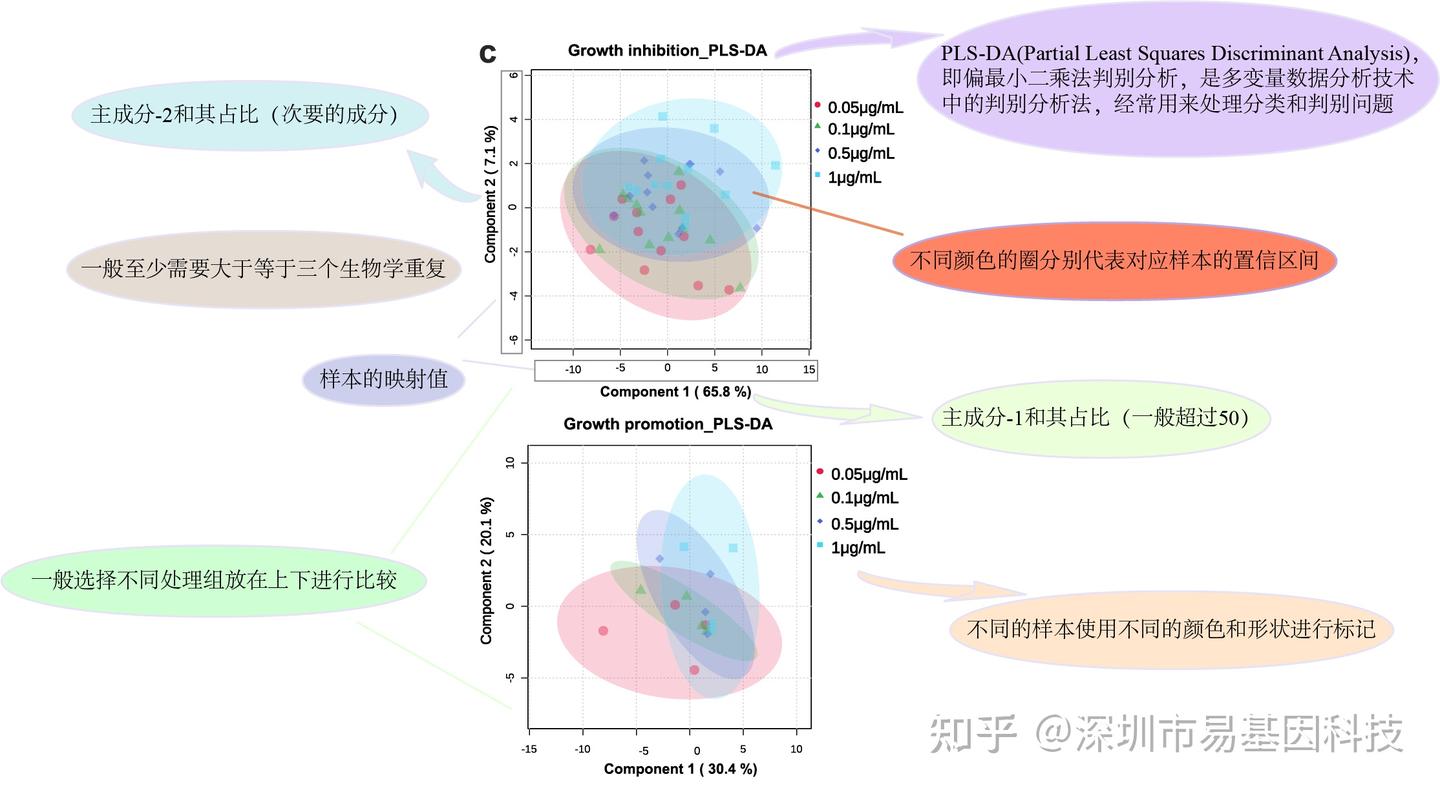
Figure 8 Pesticides elicit growth effects of gut microbiota at 0.05μg/mL, 0.1μg/mL, 0.5μg/mL, and 1μg/mL.
圖8農藥在0.05μg/mL、0.1μg/mL、0.5μg/mL和1μg/mL濃度下對腸道菌群產生生長影響。
易小結
偏最小二乘判別分析(PLS-DA)作為結合偏最小二乘回歸和判別分析的統計方法,它通過尋找最大相關性來提取最有代表的成分,進而用于預測和分類,反映在聚類圖的點與點之間的實際距離,距離越近,樣本越相似。PLS-DA在代謝組學和微生物組學等領域應用廣泛,可以有效處理共線性和高維數據。
9、tSNE
標題:Mapping the single-cell transcriptomic response of murine diabetic kidney disease to therapies(繪制小鼠糖尿病腎病單細胞轉錄組對治療反應的圖譜)
發表時間:2022年7月5日
發表期刊:Cell Metabolism(tSNE)
影響因子:IF30.9/Q1
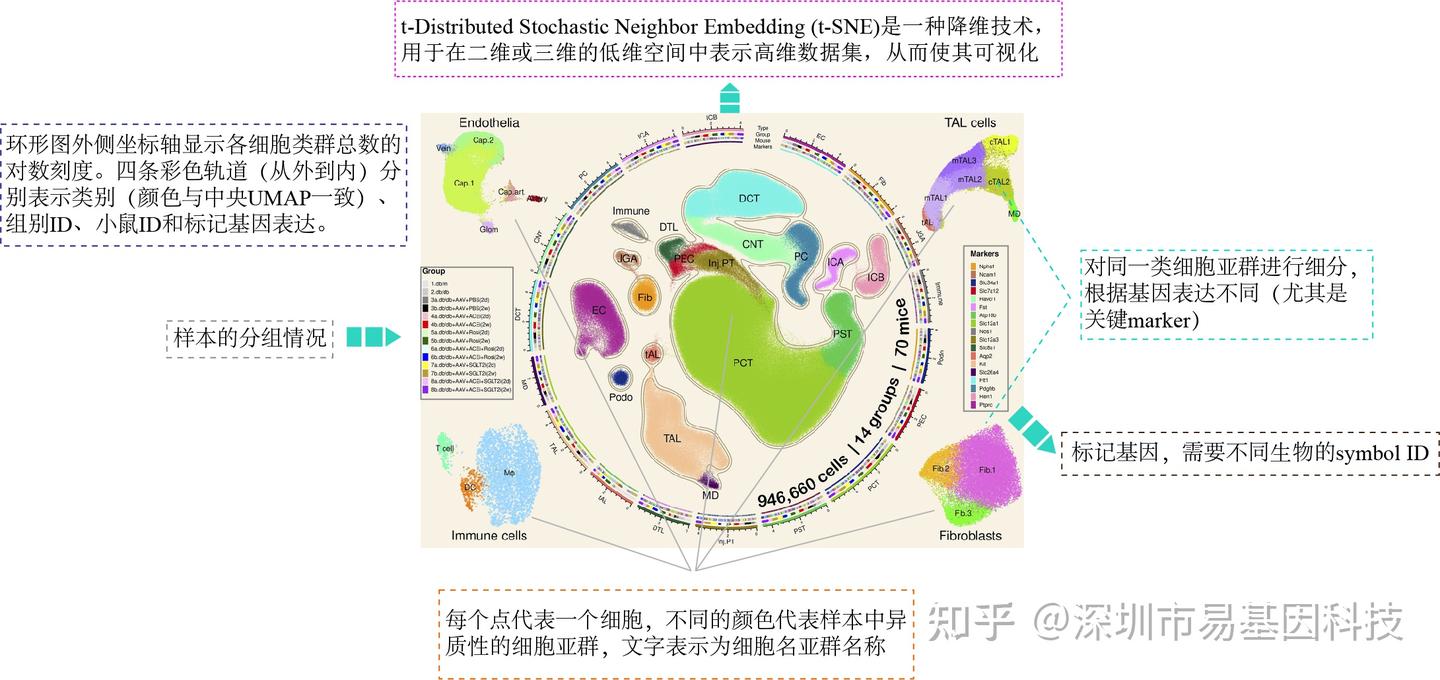
DOI:10.1016/j.cmet.2022.05.010
Figure 9 Single cell atlas of drug treatments in a mouse model of DKD.
圖9糖尿病腎病小鼠模型中藥物處理的單細胞圖譜。
易小結
TSNE(T-distributed stochastic neighbor embedding)通過發現數據集中的樣本(細胞)在原始高維空間中(細胞中)距離的概率分布,然后再去低維空間(即圖中)中重建這種概率分布。通過 t-SNE 將高維空間中的數據點嵌入到了低維空間,同時還保留了數據點在高維空間中的距離關系,常用于展示樣本之間的細胞異質性、細胞亞群圖譜、腫瘤微環境的變化、不同樣本之間的主要細胞亞群構成的變化、稀有亞群的分布等等。
10. 相關性熱圖
標題:Structure and function of the global ocean microbiome(全球海洋微生物組的結構與功能)
發表時間:2015年5月22日
發表期刊:Science(相關性熱圖)
影響因子:IF45.8/Q1
DOI:10.1126/science.1261359
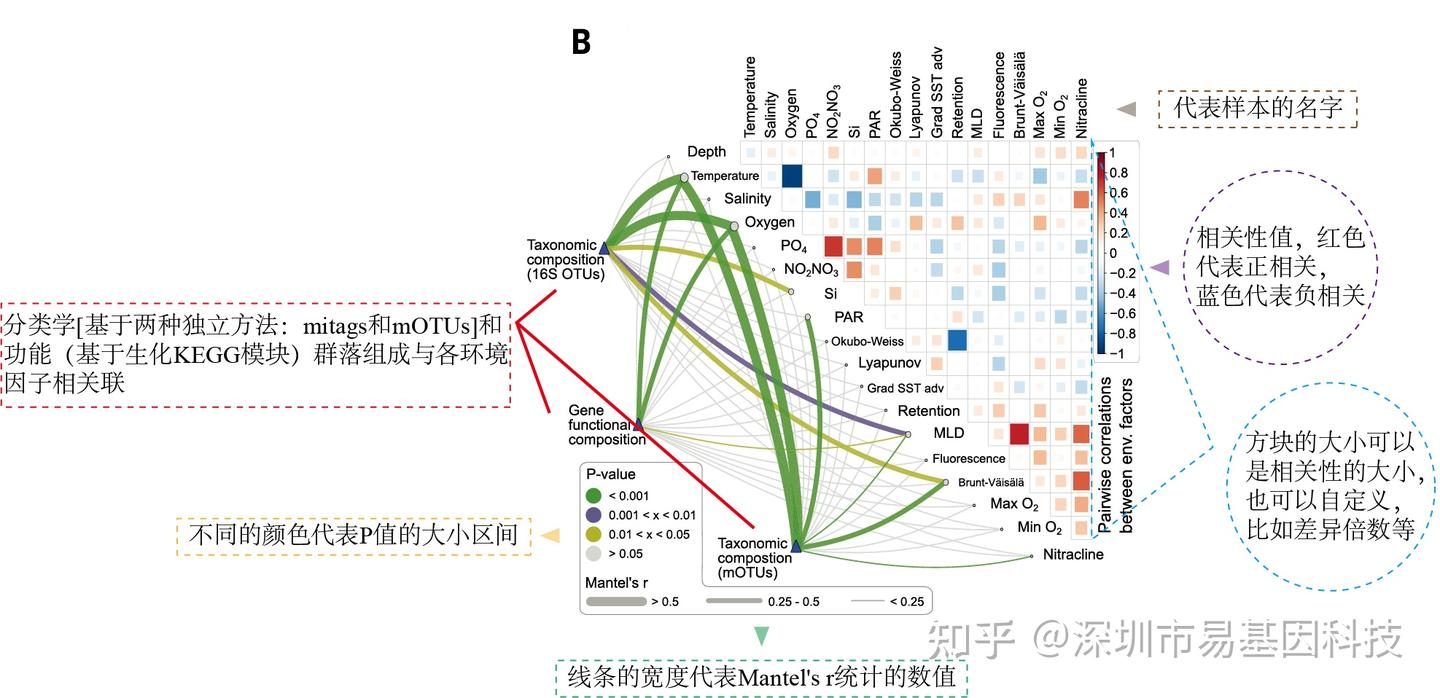
Figure 10 Pesticides elicit growth effects of gut microbiota at 0.05μg/mL, 0.1μg/mL, 0.5μg/mL, and 1μg/mL.
圖10表面微生物群落組成的環境驅動因素。
易小結
相關性熱圖通過揭示數據集中的不同樣本或者細胞之間的相關性解釋潛在的關聯性,為之后的實驗設計提供良好的基礎,廣泛應用于多組學研究和表觀遺傳、基因組、轉錄組、蛋白組、微生物組等組學研究中。
參考文獻:
1.Klomp JA, Klomp JE, Stalnecker CA, Bryant KL, Edwards AC, Drizyte-Miller K, Hibshman PS, Diehl JN, Lee YS, Morales AJ, Taylor KE, Peng S, Tran NL, Herring LE, Prevatte AW, Barker NK, Hover LD, Hallin J, Sorokin A, Kanikarla PM, Chowdhury S, Coker O, Lee HM, Goodwin CM, Gautam P, Olson P, Christensen JG, Shen JP, Kopetz S, Graves LM, Lim KH, Wang-Gillam A, Wennerberg K, Cox AD, Der CJ. Defining the KRAS- and ERK-dependent transcriptome in KRAS-mutant cancers. Science. 2024 Jun 7;384(6700):eadk0775. doi: 10.1126/science.adk0775. Epub 2024 Jun 7.
2. Mu?oz KA, Ulrich RJ, Vasan AK, Sinclair M, Wen PC, Holmes JR, Lee HY, Hung CC, Fields CJ, Tajkhorshid E, Lau GW, Hergenrother PJ. A Gram-negative-selective antibiotic that spares the gut microbiome. Nature. 2024 Jun;630(8016):429-436. doi: 10.1038/s41586-024-07502-0. Epub 2024 May 29. PMID: 38811738; PMCID: PMC12135874.
3. Han G, Sinjab A, Rahal Z, Lynch AM, Treekitkarnmongkol W, Liu Y, Serrano AG, Feng J, Liang K, Khan K, Lu W, Hernandez SD, Liu Y, Cao X, Dai E, Pei G, Hu J, Abaya C, Gomez-Bolanos LI, Peng F, Chen M, Parra ER, Cascone T, Sepesi B, Moghaddam SJ, Scheet P, Negrao MV, Heymach JV, Li M, Dubinett SM, Stevenson CS, Spira AE, Fujimoto J, Solis LM, Wistuba II, Chen J, Wang L, Kadara H. An atlas of epithelial cell states and plasticity in lung adenocarcinoma. Nature. 2024 Mar;627(8004):656-663. doi: 10.1038/s41586-024-07113-9. Epub 2024 Feb 28.
4. Bod L, Kye YC, Shi J, Torlai Triglia E, Schnell A, Fessler J, Ostrowski SM, Von-Franque MY, Kuchroo JR, Barilla RM, Zaghouani S, Christian E, Delorey TM, Mohib K, Xiao S, Slingerland N, Giuliano CJ, Ashenberg O, Li Z, Rothstein DM, Fisher DE, Rozenblatt-Rosen O, Sharpe AH, Quintana FJ, Apetoh L, Regev A, Kuchroo VK. B-cell-specific checkpoint molecules that regulate anti-tumour immunity. Nature. 2023 Jul;619(7969):348-356. doi: 10.1038/s41586-023-06231-0. Epub 2023 Jun 21.
5. Zhang R, Li G, Zhang Q, Wang Z, Xiang D, Zhang X, Chen J, Hutchins AP, Qin D, Su H, Pei D, Li D. c-JUN: a chromatin repressor that limits mesoderm differentiation in human pluripotent stem cells. Nucleic Acids Res. 2025 Jan 24;53(3):gkaf001. doi: 10.1093/nar/gkaf001. PMID: 39876710; PMCID: PMC11760979.
6. Chen J, Yang X, Cui M, Yao Z, Ouyang Z, Qu Z, Huang Y, Zhu Y, Zhao J, Chang G. Deciphering the regulatory networks of human male germline development from embryo to adulthood. Biochim Biophys Acta Mol Basis Dis. 2025 Oct;1871(7):167918. doi: 10.1016/j.bbadis.2025.167918. Epub 2025 May 21. PMID: 40409516.
7. Chen L, Yan H, Di S, Guo C, Zhang H, Zhang S, Gold A, Wang Y, Hu M, Wu D, Johnson CH, Wang X, Zhu J. Mapping pesticide-induced metabolic alterations in human gut bacteria. Nat Commun. 2025 May 10;16(1):4355. doi: 10.1038/s41467-025-59747-6. PMID: 40348778; PMCID: PMC12065874.
8. Wu H, Gonzalez Villalobos R, Yao X, Reilly D, Chen T, Rankin M, Myshkin E, Breyer MD, Humphreys BD. Mapping the single-cell transcriptomic response of murine diabetic kidney disease to therapies. Cell Metab. 2022 Jul 5;34(7):1064-1078.e6. doi: 10.1016/j.cmet.2022.05.010. Epub 2022 Jun 15. PMID: 35709763; PMCID: PMC9262852.
9. Shinichi Sunagawa et.al.,Structure and function of the global ocean microbiome. Science,1261359(2015). DOI:10.1126/science.1261359


 浙公網安備 33010602011771號
浙公網安備 33010602011771號