易基因:Plant Cell Environ:WGBS+RNA-seq助力揭示植物不定根再生的DNA甲基化調控機制|項目文章
大家好,這里是專注表觀組學十余年,領跑多組學科研服務的易基因。
植物由于不能移動,會受諸如風、雪和動物等逆境脅迫,這些脅迫可能會破壞植物結構,因此再生能力對于植物生存至關重要。近日,北京林業大學生物科學與技術學院李云教授團隊探索了植物刺槐(Robinia pseudoacacia L.)的不定根(Adventitious Roots, ARs)再生過程中DNA低甲基化(Hypomethylation)對基因調控網絡的作用。研究通過使用DNA甲基化抑制劑5-氮雜胞苷(5-azacytidine, 5-azaC)處理刺槐下胚軸切段,通過全基因組重亞硫酸鹽測序(Whole-Genome Bisulfite Sequencing, WGBS)和轉錄組測序(RNA-seq)技術,揭示了低甲基化激活以RpMYB2為中心的基因網絡,進而顯著增強不定根再生能力。相關研究成果以《DNA Hypomethylation Activates the RpMYB2-Centred Gene Network to Enhance Regeneration of Adventitious Roots》為題發表于《Plant Cell Environ》期刊。深圳易基因為本研究提供WGBS+RNA-seq技術服務助力揭示刺槐不定根再生過程中的的DNA甲基化調控機制。

標題:DNA Hypomethylation Activates the RpMYB2-Centred Gene Network to Enhance Regeneration of Adventitious Roots(DNA低甲基化激活以RpMYB2為中心的基因網絡,以增強不定根再生)
發表時間:2025年2月
發表期刊:Plant Cell Environ
影響因子:IF6.3/Q1
技術平臺:WGBS、RNA-seq(易基因金牌技術)
作者單位:北京林業大學李云等
DOI:10.1111/pce.15236

本研究利用5-azaC DNA甲基化抑制劑,使根原基的啟動和發育更早發生,從而提高刺槐的不定根再生率。WGBS揭示了在5-azaC處理樣本中,整體甲基化水平下降,而在所有背景(包括CHH、CHG和mergedCG)中,低甲基化胞嘧啶位點和區域增加,從而導致轉錄變異。酵母雙雜交(Yeast Two-Hybrid, Y2H)實驗揭示了一個以RpMYB2為中心的轉錄因子(Transcription Factors, TFs)網絡,這些轉錄因子被RpWRKY23、RpGATA23、RpSPL16以及其他基因如RpSDP、RpSS1、RpBEN1、RpGULL05和RpCUV轉錄激活,其核定位表明它們可能共定位。酵母單雜交(Yeast One-Hybrid, Y1H)實驗揭示RpMYB2互作蛋白RpGATA23、RpWRKY23與RpSK6、RpCDC48的啟動子互作,而熒光素酶報告基因實驗(Luciferase Reporting Assay, LRA)驗證了其與RpSK6結合。本研究結果表明,低甲基化介導的轉錄組修飾激活了以RpMYB2為中心的基因網絡,從而增強了刺槐下胚軸切段的不定根再生能力。這些發現為通過遺傳改良提高植物再生能力和增加木材產量,同時抵御環境損害提供了理論基礎。
易小結
本研究不僅為理解植物再生的分子機制提供了新的視角,還為通過遺傳改良提高植物再生能力和適應環境脅迫提供了潛在靶點。研究還強調了WGBS和RNA-seq技術在解析植物表觀遺傳調控中的重要作用,為未來類似研究提供了重要的技術參考。
WGBS與RNA-seq技術的聯合應用,不僅為本研究提供了從表觀遺傳修飾到基因表達調控的全方位解析,還為未來類似研究提供了一種強大的技術組合,有助于深入探索植物再生及其他復雜生物學過程中的表觀遺傳調控機制,為植物遺傳改良和分子育種提供了新的思路和潛在靶點,具有重要的科學意義和廣闊的應用前景。
WGBS植物研究更多應用案例:
項目文章|WGBS+RNA-seq揭示AtSAMS通過DNA甲基化和乙烯信號通路協同調控植物花器官發育的表觀遺傳機制
PNAS:朱健康/郎曌博/牛慶豐團隊ChIP-seq+WGBS揭示番茄果實成熟中DNA甲基化與轉錄因子互作的分子機制
Genome Biol|華中農大周道繡團隊利用BS-seq揭示水稻雄性配子發生過程DNA甲基化重編程機制
研究方法
- 植物材料收集與再生:使用刺槐種子萌發后的下胚軸切段作為實驗材料,分別在含和不含5-azaC的培養基上培養,觀察不定根再生情況。
- 石蠟切片與解剖分析:對處理和未處理的切段進行石蠟切片,觀察不定根原基的啟動和發育過程。
- WGBS分析:分析全基因組范圍內的DNA甲基化水平。
- RNA-seq分析:分析基因表達差異。
- 實時定量PCR(qRT-PCR)驗證:對RNA-seq結果中差異表達的基因進行驗證。
- 酵母雙雜交(Y2H)和酵母單雜交(Y1H)實驗:鑒定與RpMYB2互作蛋白,以及這些蛋白與下游基因啟動子的結合情況。
- 熒光素酶報告基因實驗(LRA):驗證特定轉錄因子與目標基因啟動子的結合。
- 亞細胞定位分析:通過共聚焦顯微鏡觀察關鍵蛋白在細胞中的定位。
結果圖形
(1)不定根原基的啟動與發育
研究通過石蠟切片觀察了5-azaC處理和未處理切段的不定根原基啟動和發育過程。從8日齡幼苗的下胚軸切取1cm長的切段作為外植體,并在切割后的五個時間點(包括切割后的第0天、第1天、第2天、第3天和第4天,分別標記為D0、D1、D2、D3和D4)進行橫截面分析。通過在不同時間點的組織學對比區分對照組(D1C、D2C、D3C、D4C)和5-azaC處理組(D1T、D2T、D3T、D4T)外植體中原基的發育進程以及根分生組織的增殖情況。結果顯示,5-azaC處理顯著加速了不定根原基的啟動和發育,處理組在第4天即可觀察到不定根的形成,而對照組則未能發育出完整的不定根(圖1)。這表明5-azaC通過促進細胞分裂和分化,顯著增強了不定根的再生能力。
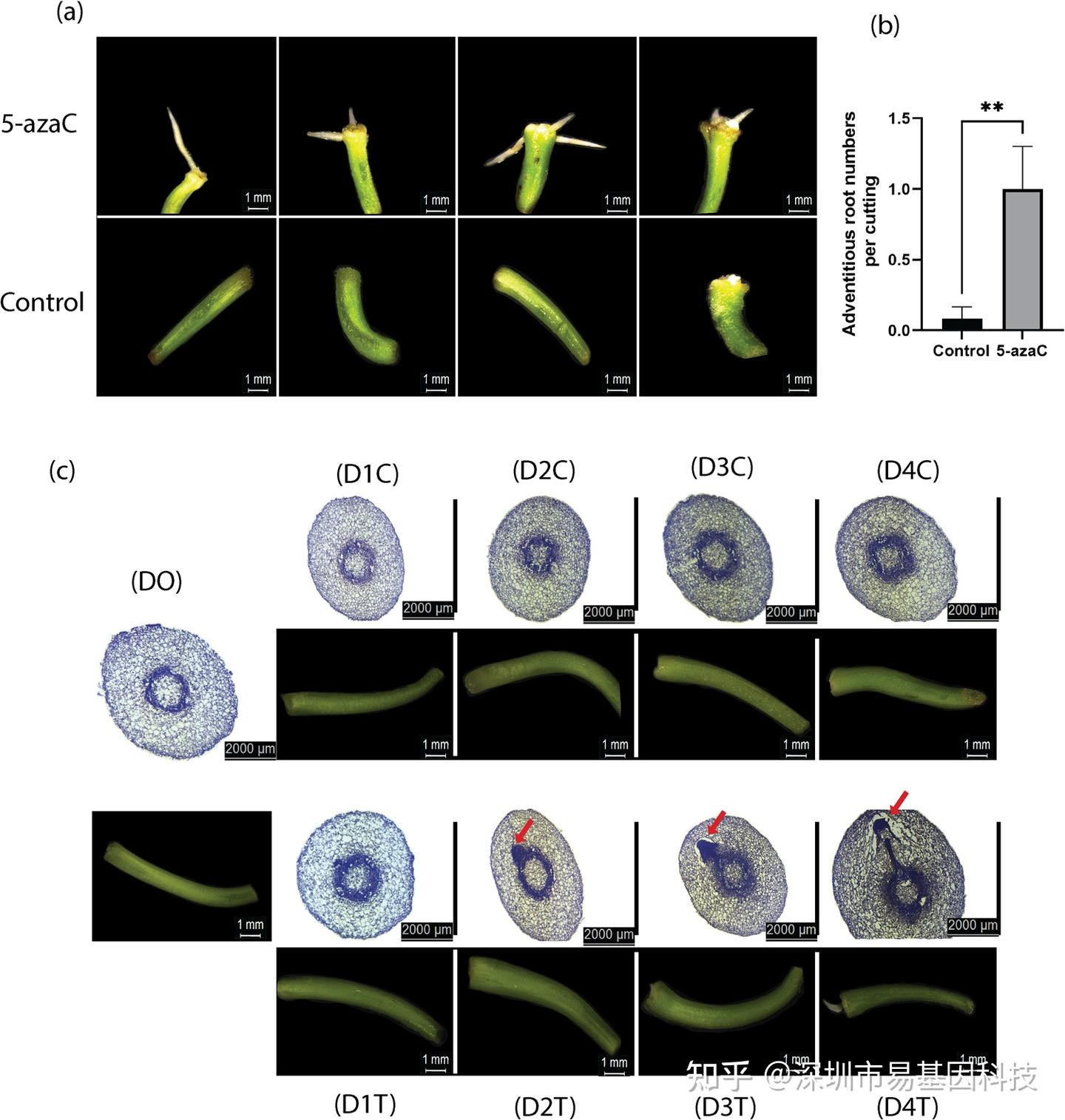
圖1:不定根形成及橫切切段的解剖學研究。
- 1cm長的下胚軸切段放置在含和不含5-azaC培養基上的形態學特征。
- 每段不定根數量。
- 下胚軸切段的解剖結構顯示了不定根原基的啟動和發育。與對照組相比,處理組(D2T)的不定根原基啟動更早。在(D3T)中形成了根冠,而在(D4T)中原基發展為功能性不定根,但在(D3C,D4C)中,根分生組織未能發育。
(2)全基因組甲基化圖譜與不定根再生能力
通過WGBS分析,研究發現5-azaC處理顯著降低了基因組整體甲基化水平,尤其是在CHH、CHG和mergedCG三種序列背景下。在基因組的不同區域(包括啟動子、基因體和終止子下游區域),處理組的甲基化水平均顯著低于對照組,尤其是在CHH背景下,甲基化水平降低更為顯著(圖2a-c)。這表明低甲基化可能通過影響基因表達調控來增強不定根的再生能力。
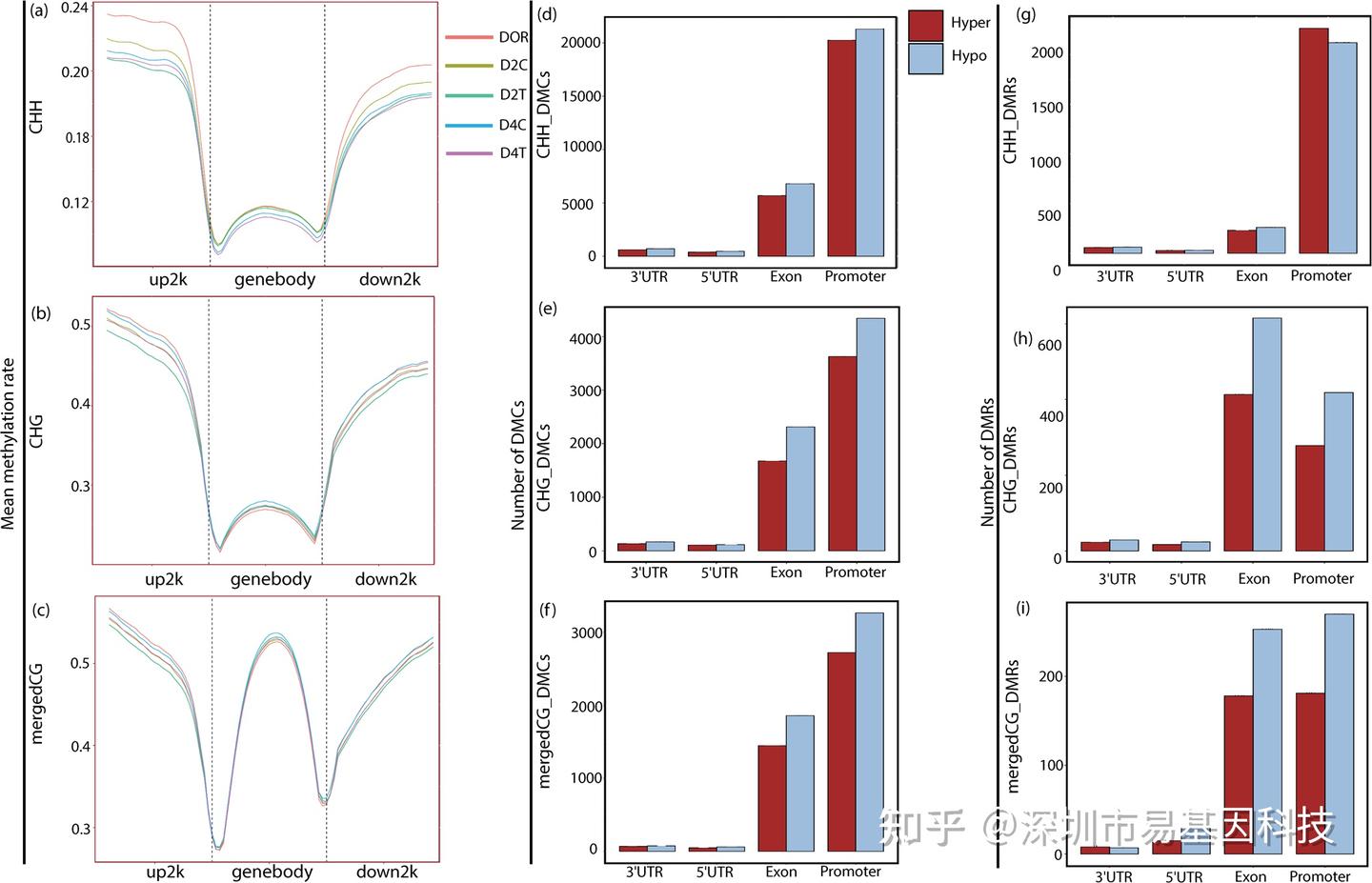
圖2:5-azaC處理介導全基因組低甲基化。
在5-azaC處理和對照樣本中,第2天和第4天的基因及其±2kb區域CHH背景(a)、CHG背景(b)和mergedCG背景(c)中的DNA甲基化水平變化。在CHH背景(d)、CGH背景(e)和mergedCG背景(f)中,不同基因組元件(包括啟動子、外顯子、5′UTR和3′UTR)中高甲基化和低甲基化差異甲基化位點(DMCs)數量。在CHH背景(g)、CGH背景(h)和mergedCG背景(i)中,不同基因組元件(包括啟動子、外顯子、5′UTR和3′UTR)中高甲基化和低甲基化差異甲基化區域(DMRs)數量。
(3)低甲基化位點和區域激活與不定根再生相關的基因
研究通過分析差異甲基化位點(DMCs)和差異甲基化區域(DMRs),發現5-azaC處理顯著增加了基因調控區域的低甲基化位點和區域數量。特別是在啟動子區域,低甲基化的DMCs和DMRs數量顯著多于高甲基化的情況(圖2d-i)。這些低甲基化位點和區域與不定根再生相關的基因表達激活密切相關,如RpMYB2、RpWRKY23等基因在低甲基化后表達顯著上調。
(4)轉錄組分析鑒定5-azaC處理激活的基因及低甲基化RpMYB2的潛在互作因子
RNA-seq分析顯示,5-azaC處理顯著改變了基因表達譜,特別是在處理4天后,差異表達基因數量顯著增加。這些差異表達基因主要包括細胞分裂、細胞分化、激素信號轉導等生物學過程(圖3)。其中,低甲基化激活的基因如RpMYB2、RpWRKY23等在不定根再生中發揮關鍵作用,且這些基因可能通過與其他基因互作來調控不定根再生。
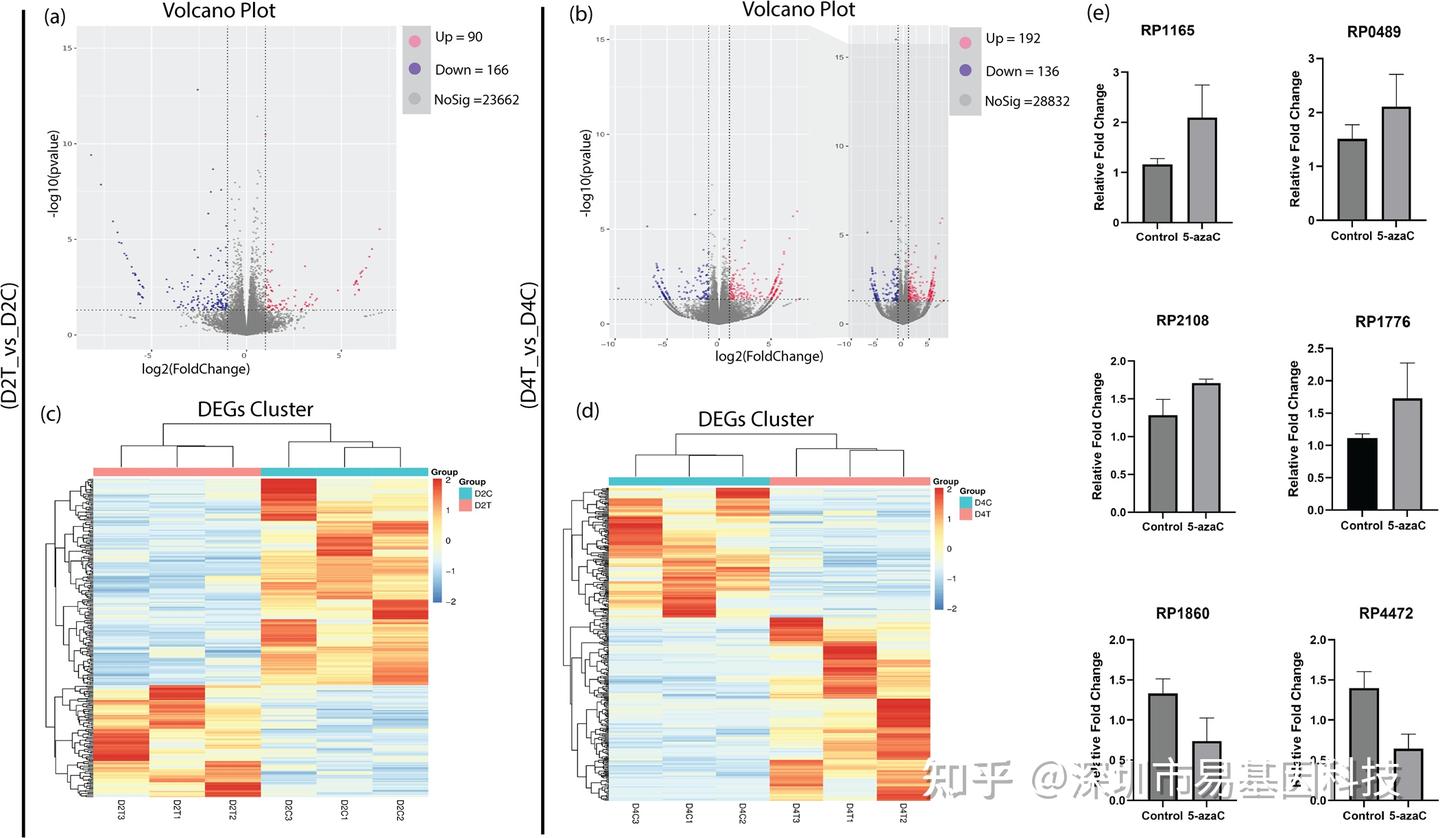
圖3:DNA低甲基化導致轉錄組變化,從而引起基因差異表達。
火山圖展示了D2C與D2T組(a)以及D4C與D4T組(b)之間的差異表達基因(DEGs)。熱圖描繪了D2C與D2T組(c)以及D4C與D4T組(d)之間的差異表達基因。所選上調和下調差異表達基因的相對表達量(e)。
(5)酵母雙雜交實驗揭示低甲基化RpMYB2在增強生根能力的基因互作網絡中的中心地位
酵母雙雜交實驗結果表明,低甲基化激活的RpMYB2是基因互作網絡的中心節點,與多個轉錄因子和功能蛋白相互作用,如RpGATA23、RpWRKY23、RpSDP等(圖4)。這些相互作用蛋白在不定根再生過程中發揮協同作用,調控細胞分裂、細胞分化和激素信號轉導等過程。

圖4:通過酵母雙雜交實驗檢測蛋白-蛋白互作。展示RpMYB2和RpSDP蛋白與其他蛋白的正向互作。
(6)酵母單雜交實驗揭示以RpMYB2為中心的基因網絡的進一步見解)
酵母單雜交實驗進一步驗證了RpMYB2互作蛋白與下游基因啟動子的結合情況。實驗發現,RpWRKY23和RpGATA23能夠與RpSK6和RpCDC48的啟動子結合,表明這些轉錄因子通過調控下游基因的表達來增強不定根的再生能力(圖5A)。
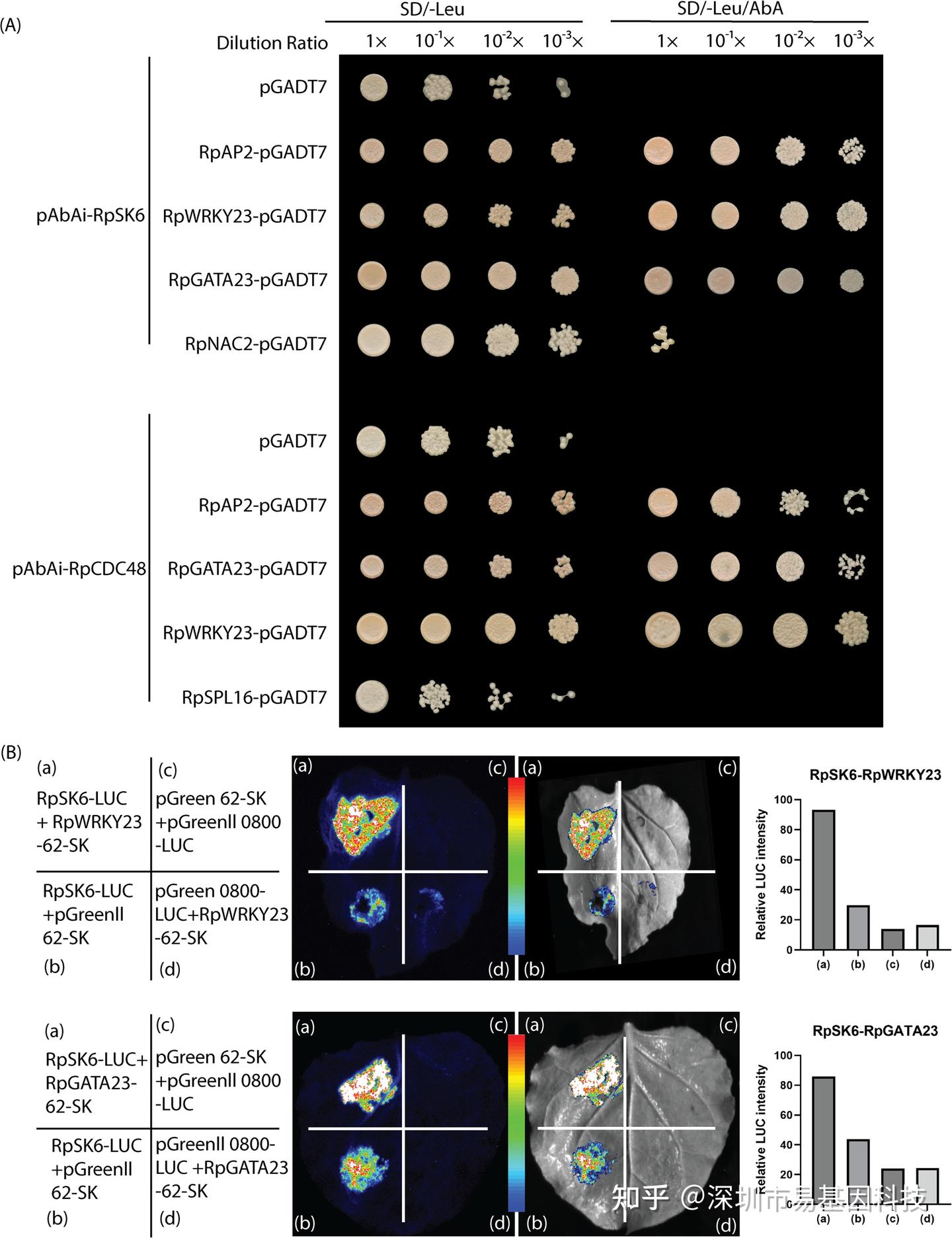
圖5:通過酵母單雜交和熒光素酶報告基因實驗分析DNA-蛋白互作。
- 酵母單雜交(Y1H)展示了不同轉錄因子與RpSK6和RpCDC48啟動子之間的相互作用。
- 熒光素酶報告基因實驗顯示轉錄因子RpWRKY23和RpGATA23與RpSK6啟動子的相互作用。
(7)熒光素酶報告基因實驗驗證RpWRKY23和RpGATA23蛋白與RpSK6啟動子的互作
熒光素酶報告基因實驗結果進一步證實了酵母單雜交實驗的發現,即RpWRKY23和RpGATA23能夠與RpSK6的啟動子結合并激活其表達(圖5B)。這表明這些轉錄因子在不定根再生過程中通過調控關鍵基因的表達發揮重要作用。
(8)亞細胞定位分析揭示以RpMYB2為中心的互作網絡中蛋白的亞細胞定位
亞細胞定位分析顯示,RpMYB2及其互作蛋白主要定位于細胞核,部分蛋白如RpSDP和RpWRKY23還定位于細胞質膜(圖6)。這表明這些蛋白在細胞核內與其他基因相互作用,調控基因表達,同時也可能參與細胞質膜相關的生物學過程。
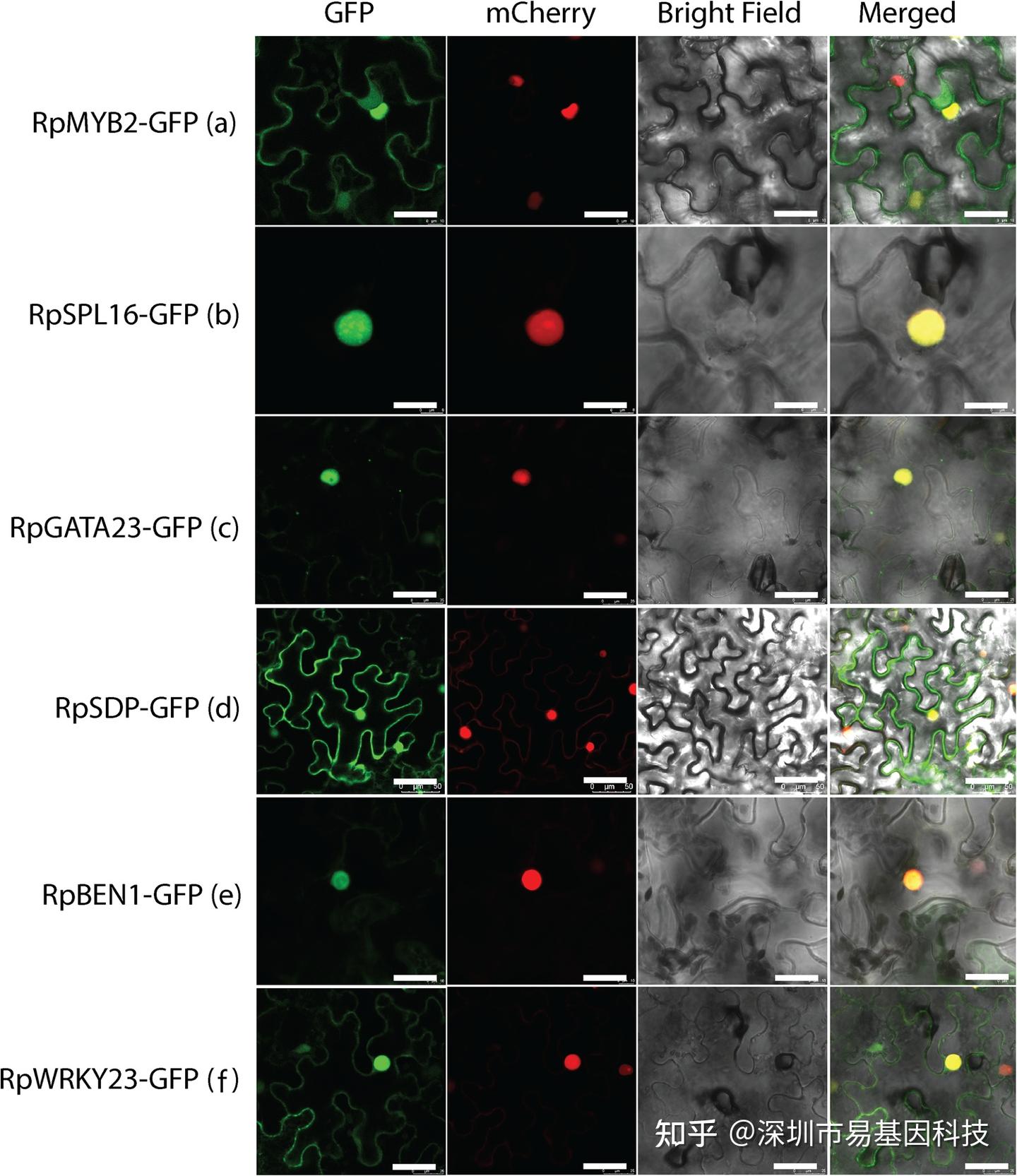
圖6:RpMYB2互作蛋白的亞細胞定位。共聚焦激光掃描顯微鏡圖像顯示了用GFP標記的蛋白的細胞內分布。所有研究的蛋白主要定位于細胞核。RpMYB2、RpSDP和RpWRKY23還顯示出在質膜定位。
結論和啟示
本研究通過WGBS和RNA-seq技術揭示了DNA低甲基化在刺槐不定根再生中的調控作用。研究發現,5-azaC處理通過降低基因組整體甲基化水平,激活了以RpMYB2為中心的基因網絡,顯著增強了不定根的再生能力。這一發現不僅為理解植物再生的分子機制提供了新的視角,還為通過遺傳改良提高植物再生能力和適應環境脅迫提供了潛在的靶點。
WGBS和RNA-seq技術在解析植物表觀遺傳調控中的重要作用
WGBS和RNA-Seq的聯合應用,研究者能夠從表觀遺傳修飾和基因表達兩個層面,全面解析DNA低甲基化在不定根再生中的調控機制。通過整合分析,研究者發現低甲基化位點和區域的變化直接導致了相關基因表達的激活,從而促進了不定根的再生。這種協同分析不僅揭示了DNA甲基化對基因表達的調控作用,還為理解植物再生過程中的表觀遺傳調控機制提供了新的視角。
關于易基因全基因組重亞硫酸鹽測序(WGBS)
全基因組重亞硫酸鹽甲基化測序(WGBS)可以在全基因組范圍內精確的檢測所有單個胞嘧啶堿基(C堿基)的甲基化水平,是DNA甲基化研究的金標準。WGBS能為基因組DNA甲基化時空特異性修飾的研究提供重要技術支持,能廣泛應用在個體發育、衰老和疾病等生命過程的機制研究中,也是各物種甲基化圖譜研究的首選方法。
易基因全基因組甲基化測序技術通過T4-DNA連接酶,在超聲波打斷基因組DNA片段的兩端連接接頭序列,連接產物通過重亞硫酸鹽處理將未甲基化修飾的胞嘧啶C轉變為尿嘧啶U,進而通過接頭序列介導的 PCR 技術將尿嘧啶U轉變為胸腺嘧啶T。
應用方向:
WGBS廣泛用于各種物種,要求全基因組掃描(不錯過關鍵位點)
- 全基因組甲基化圖譜課題
- 標志物篩選課題
- 小規模研究課題
技術優勢:
- 應用范圍廣:適用于所有參考基因組已知物種的甲基化研究;
- 全基因組覆蓋:最大限度地獲取完整的全基因組甲基化信息,精確繪制甲基化圖譜;
- 單堿基分辨率:可精確分析每一個C堿基的甲基化狀態。

易基因提供全面的表觀基因組學(DNA甲基化、DNA羥甲基化、cfDNA)和表觀轉錄組學(m6A、m5C、m1A、m7G、ac4C、RNA與蛋白互作)、DNA與蛋白互作及染色質開放性技術方案(ChIP-seq、ATAC-seq),更多表觀組學或多組學研究可關注易基因公眾號、網站、市場微信號等,期待與各位老師開展合作交流。詳詢易基因:0755-28317900。
參考文獻:
Hussain SS, Li Y, Liu J, Abbas M, Li Q, Deng H, Abbas S, Han K, Han J, Sun Y, Li Y. DNA Hypomethylation Activates the RpMYB2-Centred Gene Network to Enhance Regeneration of Adventitious Roots. Plant Cell Environ. 2024 Oct 28. doi: 10.1111/pce.15236.
相關閱讀:
1、Nature | 易基因DNA甲基化測序助力人多能干細胞向胚胎全能8細胞的人工誘導
2、Cell|易基因微量DNA甲基化測序助力中國科學家成功構建胚胎干細胞嵌合體猴,登上《細胞》封面
3、項目文章|微量WGBS+ACE-seq揭示卵巢早衰的人卵丘細胞DNA甲基化與羥甲基化表觀基因組圖譜
4、項目文章|WGBS揭示Vpr蛋白在HIV-1感染中對CD4+ T細胞DNA甲基化變化的作用
5、項目文章 | WGBS+RNA-seq揭示松材線蟲JIII階段形成過程中的DNA甲基化差異
6、項目文章 | WGBS+RNA-seq揭示黃瓜作物的“源-庫”關系受DNA甲基化調控
7、項目文章|WGBS+RNA-seq揭示AtSAMS通過DNA甲基化和乙烯信號通路協同調控植物花器官發育的表觀遺傳機制
8、項目文章|ACS Nano/IF16:WGBS及多組學研究揭示納米塑料暴露對藻類初級生產力的長期作用及表觀調控機制


 浙公網安備 33010602011771號
浙公網安備 33010602011771號